
© Getty Images/Hiroshi Watanabe
Pangenom-Projekt: Mehr Unterschiede im Erbgut als gedacht
Dank neuer Technologien kann man DNA präziser und vollständiger entschlüsseln als je zuvor. Ein neues Projekt ergründet die Diversität der Genome – und kommt zu einem überraschenden Ergebnis.
Stand:
Der emotionalste Moment kam am Ende der Pressekonferenz. „Seit zweieinhalb Jahrzehnten erforsche ich Autismus“, sagte Evan Eichler von der University of Washington School of Medicine, „aber bei 70 Prozent der Kinder können wir noch immer nicht sagen, warum sie Autismus haben.“
Einen bedeutenden Anteil jedoch werde man erklären können, wenn man über die komplette Erbinformation der Kinder verfügt und sämtliche Veränderungen darin versteht, davon ist Eichler überzeugt.
„Durch das Pangenom-Projekt kann ich in meiner verbleibenden Zeit als Forscher vielleicht dahinkommen, dass ich in zehn bis zwanzig Prozent der Fälle den Eltern erklären kann, warum ihr Kind Autismus hat – was dann hoffentlich zu besseren Behandlungsmöglichkeiten führen wird. Für mich ist das der heilige Gral in der ganzen Sache.“
Zuvor hatten Mitglieder eines internationalen Forschungskonsortiums einen weiteren Meilenstein in der Erforschung des menschlichen Erbguts vorgestellt. Das „Humane Pangenom-Projekt“ strebt an, das Genom von 350 Menschen verschiedener Ethnien in höchster Auflösung zu sequenzieren. So entsteht ein Referenzgenom, das der menschlichen Diversität ansatzweise gerecht werden soll. In eine erste Etappe sind dafür die Genome von 27 Menschen eingeflossen, die Arbeit wurde im Fachblatt „Nature“ publiziert.
Der Rest soll im kommenden Jahr folgen. Beteiligt ist auch Tobias Marschall vom Institut für Medizinische Biometrie und Bioinformatik an der Heinrich-Heine-Universität Düsseldorf, der das Projekt zusammen mit vier US-Kollegen leitet.
Neue Technologien
Nun kann man sich fragen, was der Trubel soll, nachdem 2021 bereits die Genome von 30 Millionen Menschen sequenziert worden waren und die Verbrauchsmaterialien für eine Genomsequenzierung aktuell nur noch 500 Euro kosten. Die Betonung muss auf der „höchsten Auflösung“ liegen, wie sie erst vor rund einem Jahr erreicht wurde. Im März 2022 wurde erstmals die DNA-Sequenz eines Menschen veröffentlicht, die dank verbesserter Technologie zu mehr als 99 Prozent vollständig war.
Denn die erstmals 2001 vorgestellte Sequenz eines Menschen war auch in ihrer 2003 vorgelegten „vollständigen“ Version noch gar nicht vollständig gewesen. Rund acht Prozent des Genoms waren damals unleserlich, insbesondere in den Bereichen, in denen sich größere Abschnitte wiederholen. Auch wenn nachgebessert wurde, ist die Sequenz überholt. „Diese Form einer Referenzsequenz ist ungeeignet, um die Vielfalt der menschlichen Spezies angemessen zu beschreiben, und stellt so keine gute Grundlage für die personalisierte Medizin der Zukunft dar“, sagt Tobias Marschall.
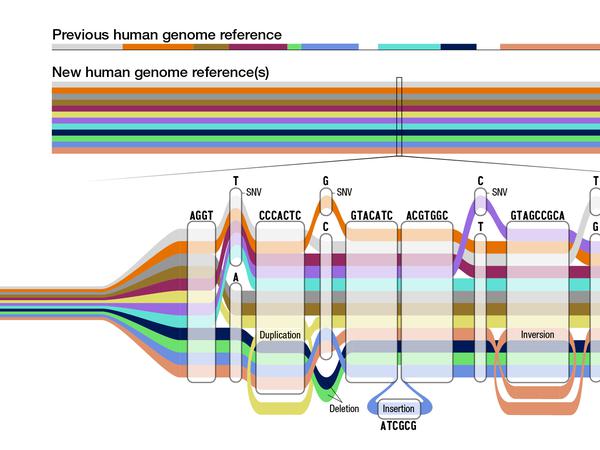
Das Pangenom, die neue Referenz für die Genomforschung, ist nicht nur vollständig, sondern auch divers. Es besteht nun nicht mehr aus einem linearen Strang aus 3,3 Milliarden Buchstaben, die für die vier Bausteine des DNA-Fadens stehen. Es gleicht eher dem U-Bahn-Netz einer Großstadt.
Manche Bereiche sind bei allen Menschen identisch oder unterscheiden sich nur durch einzelne Punktmutationen, sie verlaufen parallel. In anderen Bereichen gibt es zusätzliche Abschnitte oder auch größere Lücken, hier lassen sich die Stränge der verschiedenen Menschen nicht mehr parallel legen, sondern verlaufen eine Weile getrennt. Das könnte für Unterschiede zwischen Individuen verantwortlich sein, die sich mit den bisherigen Screening-Methoden nicht erfassen ließen.
© Darryl Leja, NHGRI
Durch das Pangenom-Projekt wird deutlich, dass die genetischen Unterschiede zwischen Menschen größer sind als zuvor angenommen „Wir finden bemerkenswerte Muster in der genetischen Variation, die wir so nicht erwartet hatten – sogar Hinweise auf neue Mechanismen der Mutation“, sagt Evan Eichler. „Bislang hatten fast alle Menschen, deren Genom wir sequenziert haben, einen anderen Satz duplizierter Gene.“
Diagnose von Behinderungen
Hinweise auf die Bedeutung solcher strukturellen Unterschiede gibt es schon länger. „Wenn Kinder in Deutschland mit Behinderungen auf die Welt kommen, dann suchen wir vor allem nach Doppelungen oder auch Verlusten von größeren Abschnitten im Genom“, sagt André Reis, Direktor des Humangenetischen Instituts am Universitätsklinikum Erlangen, der an dem Pangenom-Projekt nicht beteiligt ist. Denn wenn ein Genom sich durch einen längeren Abschnitt von anderen unterscheide, dann wirke sich das meist auch stärker aus als vertauschte Einzelbausteine.
Das ganze Genom behinderter Kinder wird in Deutschland in der Regel nicht sequenziert, auch nicht mit der unvollständigen, relativ kostengünstigen und weltweit millionenfach angewandten Methode. Die Krankenkassen übernehmen bislang nur die Kosten von 2800 Euro, für die man zwei Prozent eines Genoms entschlüsseln kann – den Bereich, der die eigentlichen Gene enthält. Das kann schon helfen, eine Diagnose zu erhalten. „Auch wenn es oft keine Heilung gibt, wissen alle Beteiligten dann zumindest, was auf sie zukommt“, sagt Reis.
Entsprechend erwartet der Humangenetiker auch nicht, dass sich das Pangenom-Projekt mit seiner 99-Prozent-Sequenzierung derzeit auf die klinische Praxis auswirken wird – wohl aber, dass es die Forschung voranbringt. Mit der neuen, hochkomplexen Datenfülle können vorerst nur Spezialisten etwas anfangen. Solche wie Evan Eichler, der die Entstehung von Autismus verstehen will.
- showPaywall:
- false
- isSubscriber:
- false
- isPaid: